13 Symulacje
library('gstat')
library('sp')
library('ggplot2')
library('raster')
library('geostatbook')
data(punkty)
data(siatka)13.1 Symulacje geostatystyczne
13.1.1 Symulacje geostatystyczne
Kriging daje optymalne predykcje, czyli wyznacza najbardziej potencjalnie możliwą wartość dla wybranej lokalizacji. Dodatkowo, efektem krigingu jest wygładzony obraz. W konsekwencji wyniki estymacji różnią się od danych pomiarowych. Uzyskiwana jest tylko (aż?) predykcja, a prawdziwa wartość jest niepewna… Korzystając z symulacji geostatystycznych nie tworzymy predykcji, ale generujemy równie prawdopodobne możliwości poprzez symulację z rozkładu prawdopodobieństwa (wykorzystując genereator liczb losowych).
13.1.2 Symulacje geostatystyczne
Właściwości symulacji geostatystycznych:
- Efekt symulacji ma bardziej realistyczny przestrzenny wzór (ang. pattern) niż kriging, którego efektem jest wygładzona reprezentacja rzeczywistości
- Każda z symulowanych map jest równie prawdopodobna
- Symulacje pozwalają na przedstawianie niepewności interpolacji
- Jednocześnie - kriging jest znacznie lepszy, gdy naszym celem jest jak najdokładniejsza predykcja
13.2 Typy symulacji
13.2.1 Typy symulacji
Istnieją dwa typy symulacji geostatystycznych:
- Symulacje bezwarunkowe (ang. Unconditional Simulations) - wykorzystujące semiwariogram, żeby włączyć informację przestrzenną, ale wartości ze zmierzonych punktów nie są w niej wykorzystywane
- Symulacje warunkowe (ang. Conditional Simulations) - opiera się ona o średnią wartość, strukturę kowariancji oraz obserwowane wartości
13.3 Symulacje bezwarunkowe
Symulacje bezwarunkowe w pakiecie gstat tworzy się z wykorzystaniem funkcji krige(). Podobnie jak w przypadku estymacji geostatystycznych, należy tutaj podać wzór, model, siatkę, średnią globalną (beta), oraz liczbę sąsiednich punktów używanych do symulacji (w przykadzie poniżej nmax=30). Należy wprowadzić również informacje, że nie korzystamy z danych punktowych (locations=NULL) oraz że chcemy stworzyć dane sztuczne (dummy=TRUE). Ostani argument (nsim=4) informuje o liczbie symulacji do przeprowadzenia.
sym_bezw1 <- krige(formula=z~1, model=vgm(psill=0.025, model='Exp', range=100), newdata=siatka, beta=1,
nmax=30, locations=NULL, dummy=TRUE, nsim=4)## [using unconditional Gaussian simulation]spplot(sym_bezw1, main='Przestrzennie skorelowana powierzchnia \nśrednia=1,
sill=0.025, zasięg=100, model wykładniczy')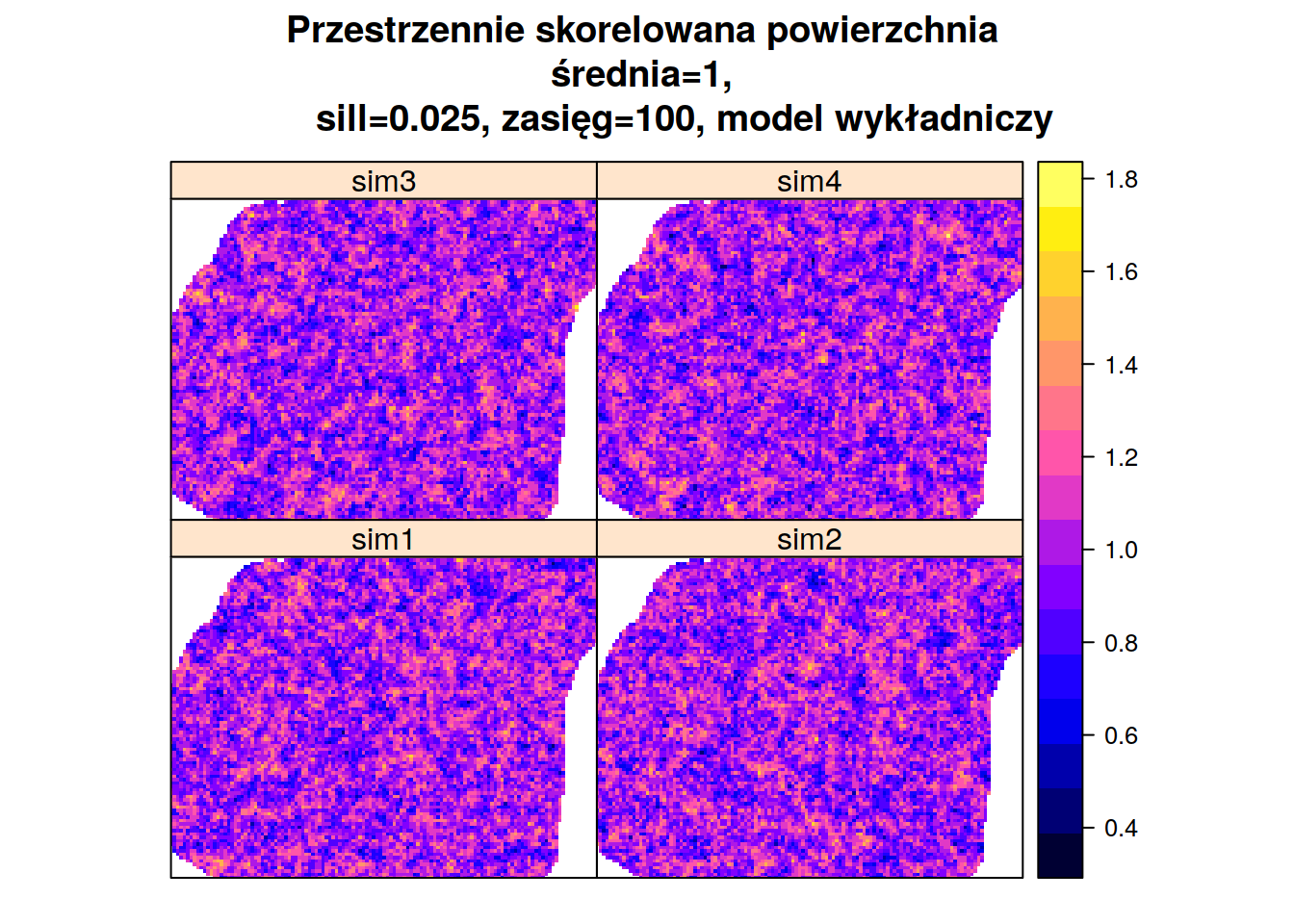
sym_bezw2 <- krige(formula=z~1, , model=vgm(psill=0.025, model='Exp', range=1500), newdata=siatka, beta=1,
nmax=30, locations=NULL, dummy=TRUE, nsim=4)## [using unconditional Gaussian simulation]spplot(sym_bezw2, main='Przestrzennie skorelowana powierzchnia \nśrednia=1,
sill=0.025, zasięg=1500, model wykładniczy')
13.4 Symulacje warunkowe
13.4.1 Sekwencyjna symulacja gaussowska (ang. Sequential Gaussian simulation)
Jednym z podstawowych typów symulacji warunkowych jest sekwencyjna symulacja gaussowska. Polega ona na:
- Wybraniu lokalizacji nie posiadającej zmierzonej wartości badanej zmiennej
- Krigingu wartości tej lokalizacji korzystając z dostępnych danych, co pozwala na uzyskanie rozkładu prawdopodobieństwa badanej zmiennej
- Wylosowaniu wartości z rozkładu prawdopodobieństwa za pomocą generatora liczba losowych i przypisaniu tej wartości do lokalizacji
- Dodaniu symulowanej wartości do zbioru danych i przejściu do kolejnej lokalizacji
- Powtórzeniu poprzednich kroków, aż do momentu gdy nie pozostanie już żadna nieokreślona lokalizacja
Sekwencyjna symulacja gaussowska wymaga zmiennej posiadającej wartości o rozkładzie zbliżonym do normalnego. Można to sprawdzić poprzez wizualizacje danych (histogram, wykres kwantyl-kwantyl) lub też test statystyczny (test Shapiro-Wilka). Zmienna temp nie ma rozkładu zbliżonego do normalnego.
ggplot(punkty@data, aes(temp)) + geom_histogram()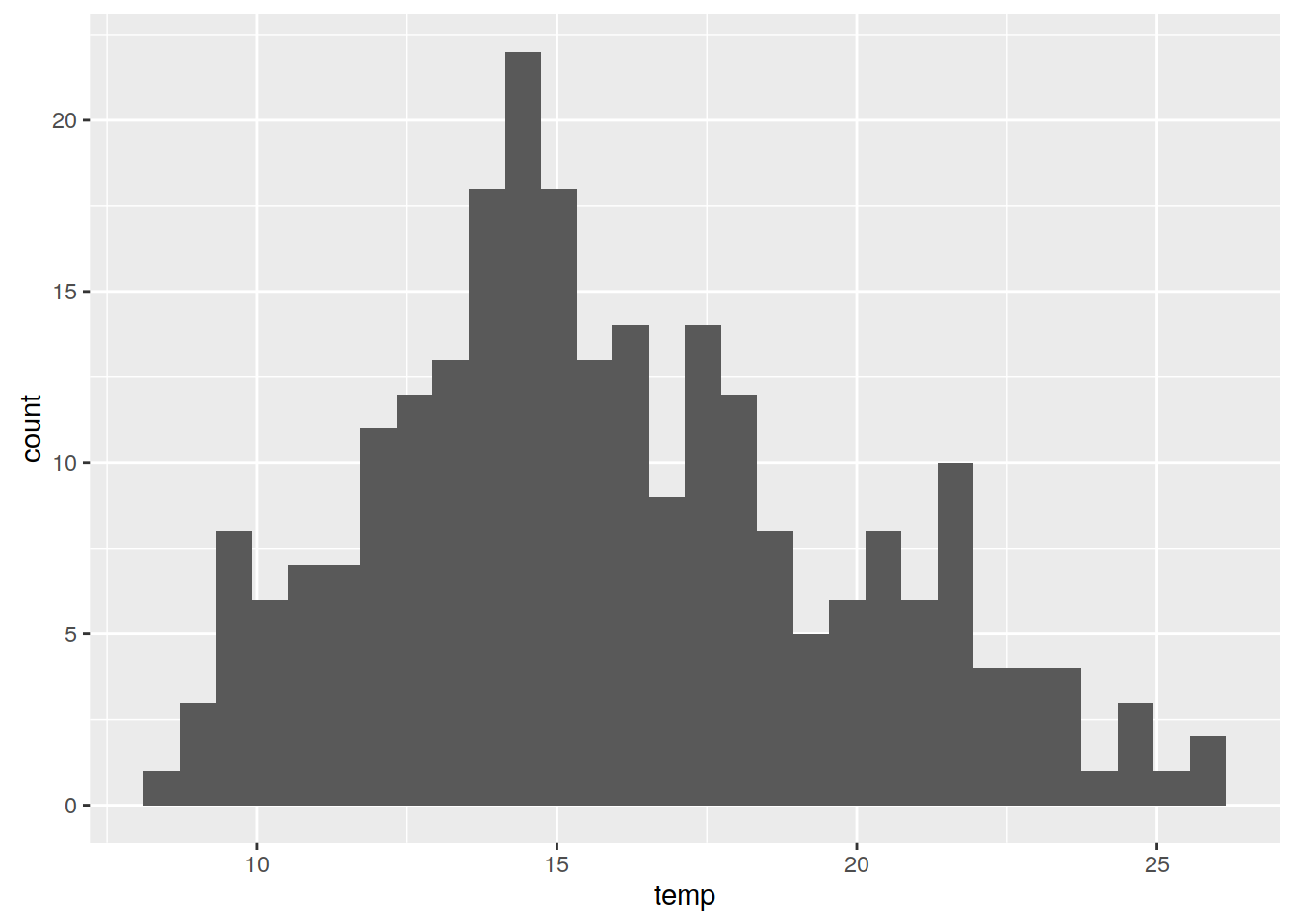
ggplot(punkty@data, aes(sample=temp)) + stat_qq()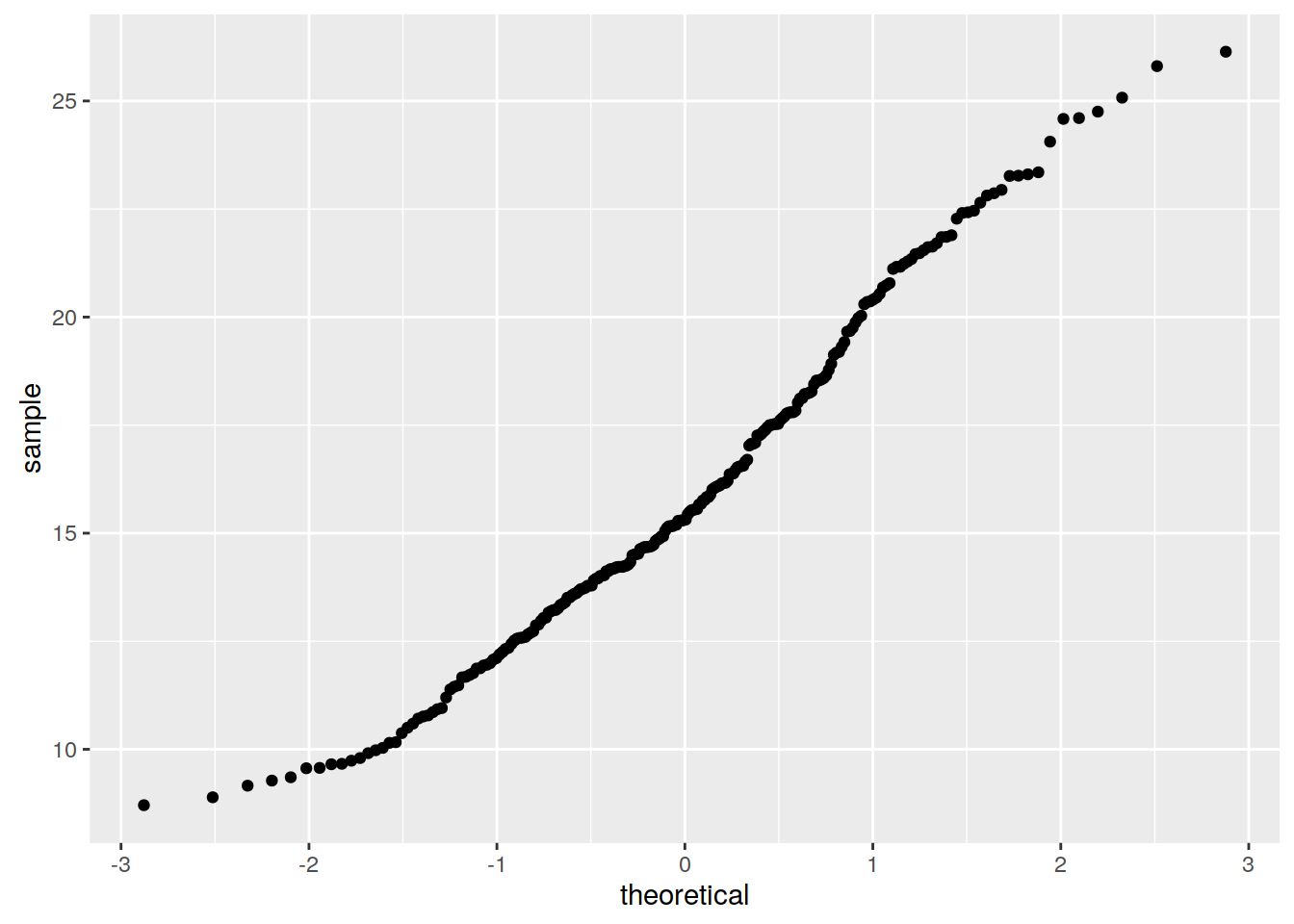
shapiro.test(punkty$temp)##
## Shapiro-Wilk normality test
##
## data: punkty$temp
## W = 0.97683, p-value = 0.0004194Na potrzeby symulacji zmienna temp została zlogarytmizowna.
punkty$temp_log <- log(punkty$temp)
ggplot(punkty@data, aes(temp_log)) + geom_histogram()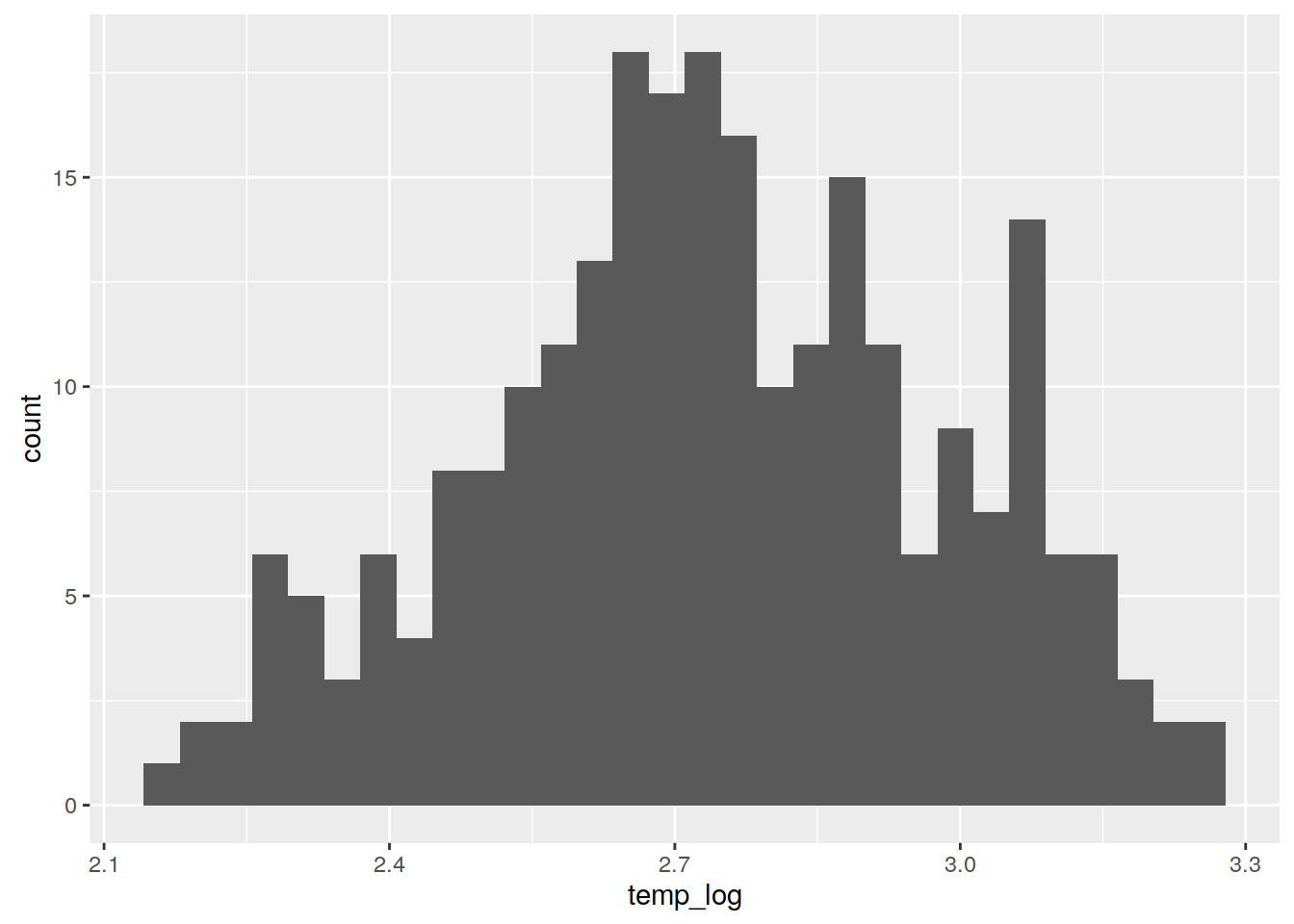
ggplot(punkty@data, aes(sample=temp_log)) + stat_qq()
shapiro.test(punkty$temp_log)##
## Shapiro-Wilk normality test
##
## data: punkty$temp_log
## W = 0.98907, p-value = 0.05568Dalsze etapy przypominają przeprowadzenie estymacji statystycznej, jedynym wyjątkiem jest dodanie argumentu mówiącego o liczbie symulacji do przeprowadzenia (nsim w funkcji krige()).
vario <- variogram(temp_log~1, punkty)
model <- vgm(0.05, model = 'Sph', range = 4500, nugget=0.005)
fitted <- fit.variogram(vario, model)
plot(vario, model=fitted)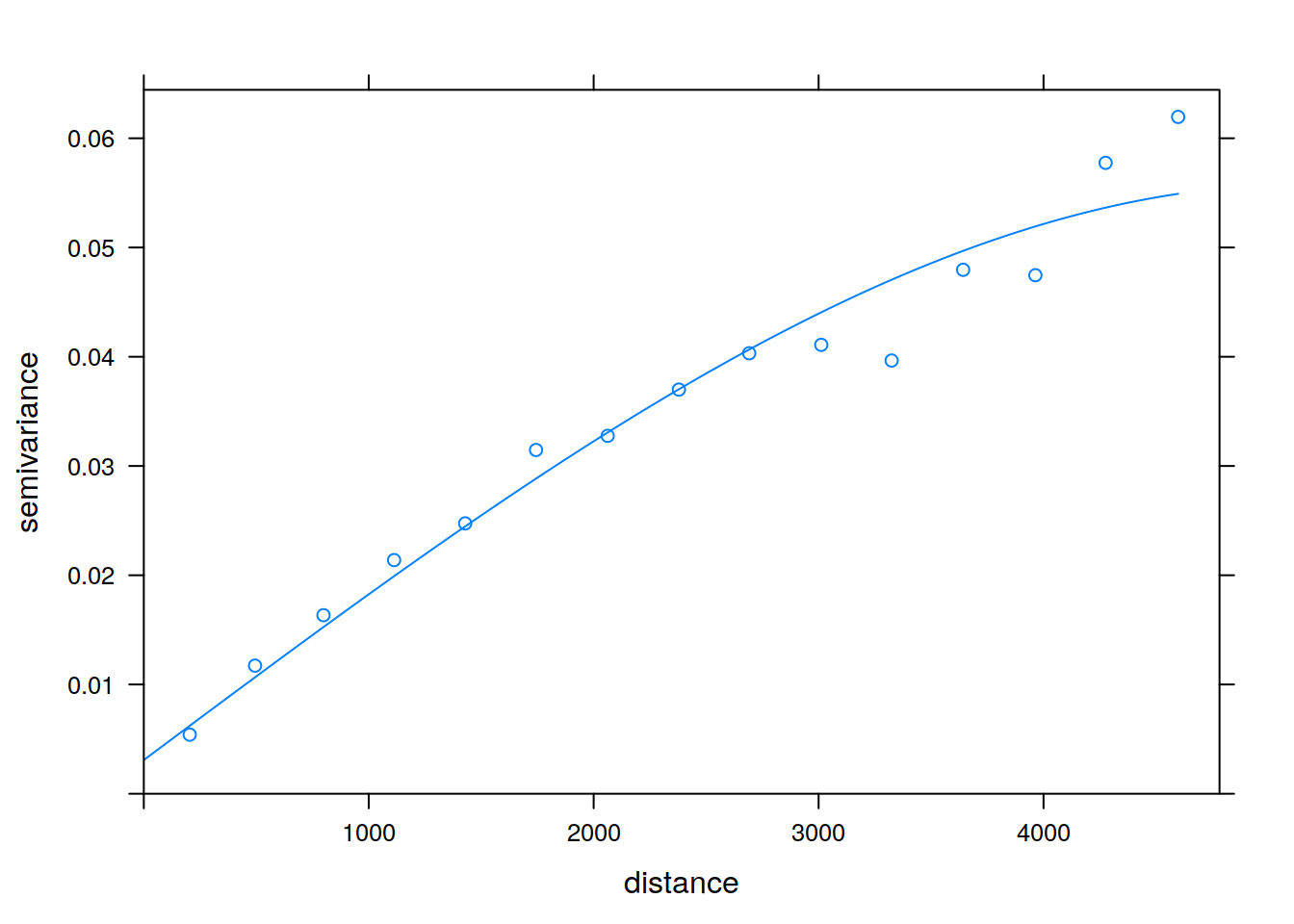
sym_ok <- krige(temp_log~1, punkty, siatka, model=fitted, nmax=30, nsim=4)## drawing 4 GLS realisations of beta...
## [using conditional Gaussian simulation]spplot(sym_ok)
Wyniki symulacji można przetworzyć do pierwotnej jednostki z użyciem funkcji wykładniczej (exp).
summary(sym_ok)## Object of class SpatialPixelsDataFrame
## Coordinates:
## min max
## x 745586.7 756926.7
## y 712661.2 721211.2
## Is projected: TRUE
## proj4string :
## [+init=epsg:2180 +proj=tmerc +lat_0=0 +lon_0=19 +k=0.9993 +x_0=500000
## +y_0=-5300000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs]
## Number of points: 10993
## Grid attributes:
## cellcentre.offset cellsize cells.dim
## s1 745586.7 90 127
## s2 712661.2 90 96
## Data attributes:
## sim1 sim2 sim3 sim4
## Min. :2.008 Min. :2.067 Min. :2.008 Min. :2.014
## 1st Qu.:2.548 1st Qu.:2.563 1st Qu.:2.567 1st Qu.:2.548
## Median :2.710 Median :2.730 Median :2.730 Median :2.715
## Mean :2.729 Mean :2.740 Mean :2.734 Mean :2.730
## 3rd Qu.:2.915 3rd Qu.:2.919 3rd Qu.:2.894 3rd Qu.:2.912
## Max. :3.425 Max. :3.506 Max. :3.361 Max. :3.525sym_ok@data <- as.data.frame(apply(sym_ok@data, 2, exp))
summary(sym_ok)## Object of class SpatialPixelsDataFrame
## Coordinates:
## min max
## x 745586.7 756926.7
## y 712661.2 721211.2
## Is projected: TRUE
## proj4string :
## [+init=epsg:2180 +proj=tmerc +lat_0=0 +lon_0=19 +k=0.9993 +x_0=500000
## +y_0=-5300000 +ellps=GRS80 +towgs84=0,0,0,0,0,0,0 +units=m +no_defs]
## Number of points: 10993
## Grid attributes:
## cellcentre.offset cellsize cells.dim
## s1 745586.7 90 127
## s2 712661.2 90 96
## Data attributes:
## sim1 sim2 sim3 sim4
## Min. : 7.445 Min. : 7.902 Min. : 7.45 Min. : 7.493
## 1st Qu.:12.787 1st Qu.:12.973 1st Qu.:13.02 1st Qu.:12.785
## Median :15.035 Median :15.329 Median :15.33 Median :15.107
## Mean :15.814 Mean :15.961 Mean :15.83 Mean :15.819
## 3rd Qu.:18.443 3rd Qu.:18.520 3rd Qu.:18.07 3rd Qu.:18.392
## Max. :30.714 Max. :33.311 Max. :28.82 Max. :33.940spplot(sym_ok)
Symulacje geostatystyczne pozwalają również na przedstawianie niepewności interpolacji. W tym wypadku należy wykonać znacznie więcej powtórzeń (np. nsim=100). Uzyskane wyniki należy przeliczyć do oryginalnej jednostki, a następnie wyliczyć odchylenie standardowe ich wartości (używając funkcji stack() i calc() z pakietu raster).
sym_sk <- krige(temp_log~1, punkty, siatka, model=fitted, beta=2.7, nsim=100, nmax=30)## [using conditional Gaussian simulation]sym_sk@data <- as.data.frame(apply(sym_sk@data, 2, exp))
sym_sk <- stack(sym_sk)
sym_sk_sd <- calc(sym_sk, fun = sd)W efekcie otrzymujemy mapę odchylenia standardowego symulowanych wartości. Można na niej odczytać obszary o najpewniejszych (najmniej zmiennych) wartościach (niebieski kolor) oraz obszary o największej zmienności cechy (kolor żółty).
spplot(sym_sk_sd)
13.5 Sekwencyjna symulacja danych kodowanych
13.5.1 Sekwencyjna symulacja danych kodowanych (ang. Sequential indicator simulation)
Symulacje geostatystyczne można również zastosować do danych binarnych. Dla potrzeb przykładu tworzona jest nowa zmienna temp_ind przyjmująca wartość TRUE dla pomiarów o wartościach temperatury niższych niż 12 stopni Celsjusza oraz FALSE dla pomiarów o wartościach temperatury równych lub wyższych niż 12 stopni Celsjusza.
summary(punkty$temp) ## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 8.706 13.280 15.310 15.950 18.270 26.140punkty$temp_ind <- punkty$temp < 12
summary(punkty$temp_ind) ## Mode FALSE TRUE NA's
## logical 212 38 0W tej metodzie kolejne etapy przypominają przeprowadzenie krigingu danych kodowanych. Jedynie w funkcji krige() należy dodać argument mówiący o liczbie symulacji do przeprowadzenia (nsim ).
vario_ind <- variogram(temp_ind~1, punkty)
plot(vario_ind)
model_ind <- vgm(0.14, model = 'Sph', range = 2000, nugget = 0.02)
plot(vario_ind, model=model_ind)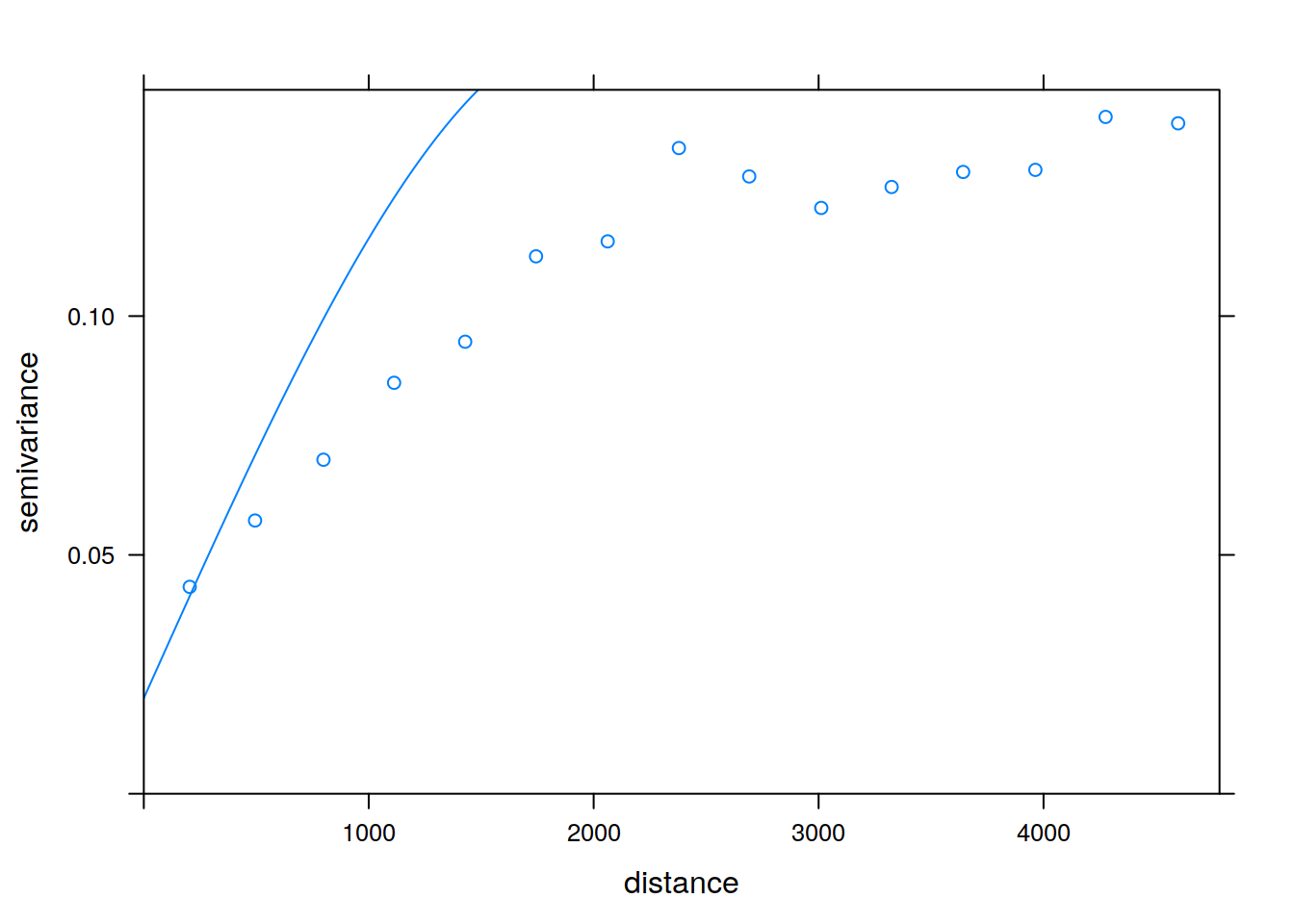
fitted_ind <- fit.variogram(vario_ind, model_ind)
fitted_ind## model psill range
## 1 Nug 0.03299461 0.000
## 2 Sph 0.09866551 3006.403plot(vario_ind, model=fitted_ind)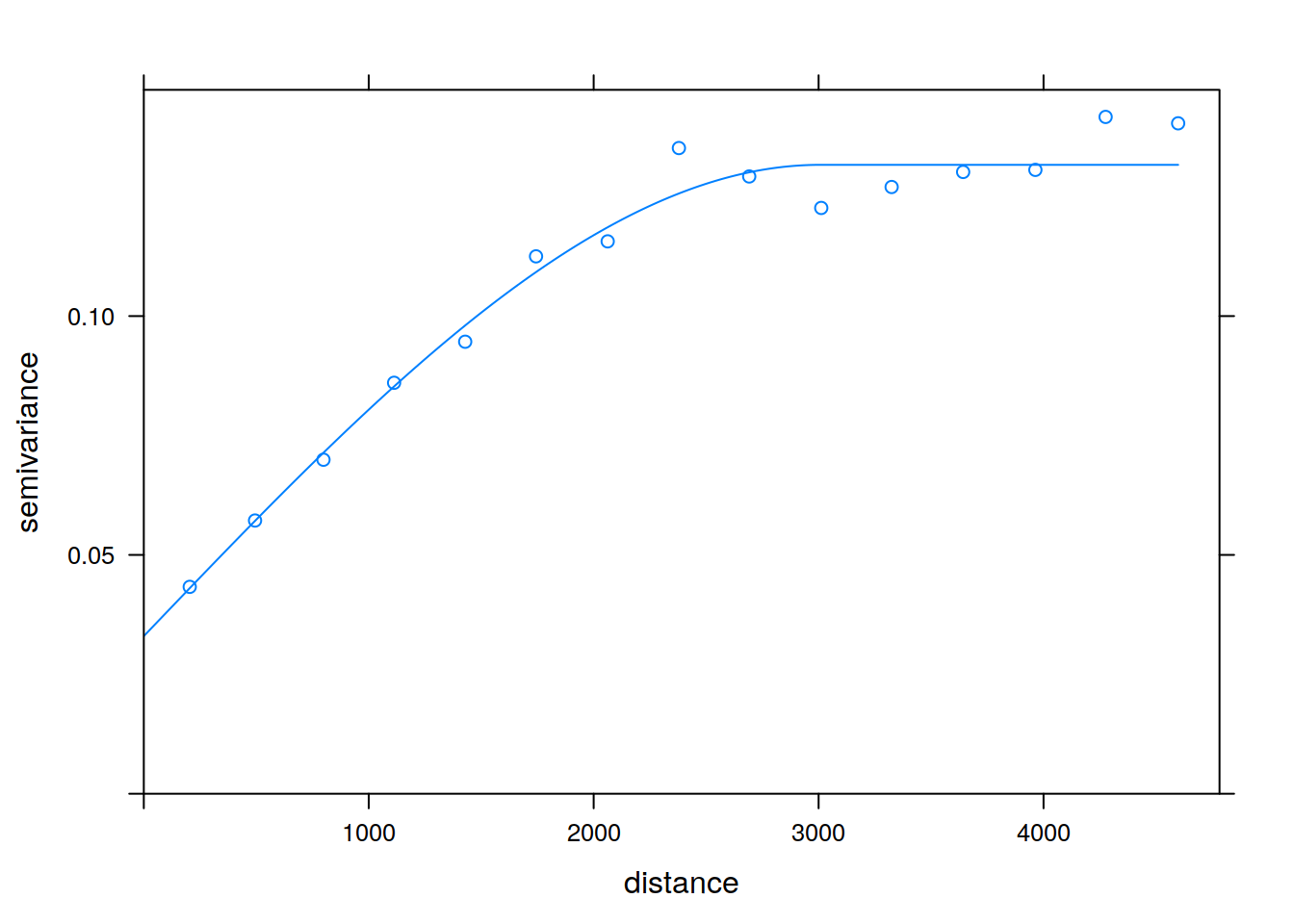
sym_ind <- krige(temp_ind~1, punkty, siatka, model=fitted_ind, indicators=TRUE, nsim=4, nmax=30)## drawing 4 GLS realisations of beta...
## [using conditional indicator simulation]Wynik symulacji danych kodowanych znacząco różni się od wyniku krigingu danych kodowanych. W przeciwieństwie do tej drugiej metody, w rezultacie symulacji nie otrzymujemy prawdopodobieństwa zajścia danej klasy, ale konkretne wartości 1 lub 0.
spplot(sym_ind, main='Symulacje warunkowe')